La nueva perspectiva molecular del gen en la era posgenómica
DOI:
https://doi.org/10.54502/msuceva.v2n1a7Palabras clave:
Dogma central de la Biología Molecular, gen, genética, genómica, posgenómicaResumen
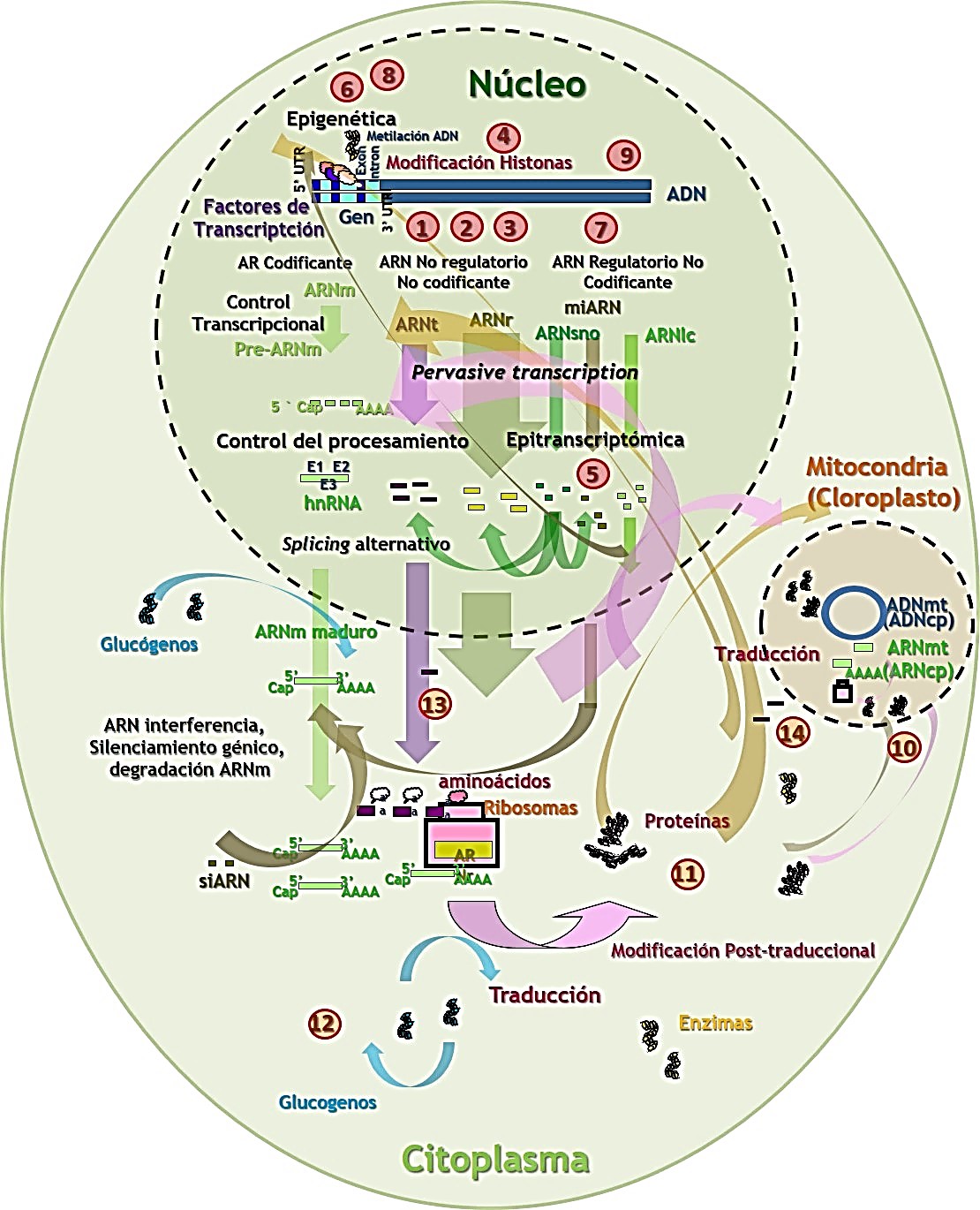
El Proyecto ENCODE (Encyclopedia of DNA Elements) fue considerado como una continuación del Proyecto Genoma Humano (PGH) que tenía como objetivo identificar todos los elementos funcionales en el genoma y profundizar en el análisis de la expresión del gen y su complejidad. A pesar de los cientos de miles de proteínas presentes en el ser humano únicamente 20.000 genes habían sido descritos. El objetivo principal del proyecto ENCODE era determinar el papel del resto del componente del genoma, excluyendo las regiones codificantes o genes. Sin embargo, partir de ENCODE, en la nueva era posgenómica, se evidenciaron nuevos fenómenos moleculares relacionados con el genoma y localizados en el núcleo de la célula (incluyendo las variaciones de copia del genoma, los genes de fusión, los fenómenos de pleiotropía, la herencia epigenética, la epitranscriptómica, las epimutaciones, los daños del ADN, la transmisión transgeneracional de información ambiental o la agrupación del ADN en una cuádruple hélice) o no relacionados con el genoma y localizados en el citoplasma celular (incluyendo la herencia mediada por material extra-genómico, las modificaciones postraduccionales de proteínas, la presencia de glucógenos y la regulación de ARNt nuclear, cloroplástico y mitocondrial) que cuestionan el concepto de gen y el Dogma Central de la Biología Molecular (DCBM). Estos nuevos fenómenos que discutiremos a continuación han supuesto una nueva perspectiva molecular del gen y del DCBM.
Descargas
Métricas
Citas
Sanger F, Coulson AR. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. Journal of Molecular Biology 1975;94:441–8. https://doi.org/10.1016/0022-2836(75)90213-2 DOI: https://doi.org/10.1016/0022-2836(75)90213-2
García-Sancho M. A New Insight into Sanger’s Development of Sequencing: From Proteins to DNA, 1943–1977. Journal of the History of Biology 2010;43:265–323. https://doi.org/10.1007/s10739-009-9184-1 DOI: https://doi.org/10.1007/s10739-009-9184-1
National Human Genome Research Institute (NIH). MINC Toolkit. The Forefront of Genomics 2022. https://www.genome.gov/minc.
Brown TA. Genomes 4. Other Titles: Genomes | Genomes Four Description: 4th. | New York, NY : Garland Science, [2017] | Preceded By: Garland Science; 2018. https://doi.org/10.1201/9781315226828. DOI: https://doi.org/10.1201/9781315226828
Ronaghi M, Karamohamed S, Pettersson B, Uhlén M, Nyrén P. Real-time DNA sequencing using detection of pyrophosphate release. Analytical Biochemistry 1996;242:84–9. https://doi.org/10.1006/abio.1996.0432 DOI: https://doi.org/10.1006/abio.1996.0432
Illumina. Powering the heroes on the front lines with sequencing solutions to address a pandemic. Illumina Innovation Technologies 2022. https://www.illumina.com/.
Thermo Fisher Scientific. Featured education & support 2022. https://www.thermofisher.com/co/en/home.html.
PacBio. Sequence with confidence. Your Partner in an Evolving Pandemic 2022. https://www.pacb.com/.
MGI. High speed, high flexibility, and ultra-high throughput turbocharge your sequencing. DNBSEQ-T7 Brochure 2022. https://en.mgi-tech.com/products/instruments_info/5/.
Callebaut W. Scientific perspectivism: A philosopher of science’s response to the challenge of big data biology. Studies in History and Philosophy of Science Part C: Studies in History and Philosophy of Biological and Biomedical Sciences 2012;43:69–80.
https://doi.org/10.1016/j.shpsc.2011.10.007 DOI: https://doi.org/10.1016/j.shpsc.2011.10.007
Selzer PM, Marhöfer RJ, Koch O. Applied Bioinformatics. Cham: Springer International Publishing; 2018. https://doi.org/10.1007/978-3-319-68301-0. DOI: https://doi.org/10.1007/978-3-319-68301-0
National Library of Medicine (NIH). Welcome to NCBI. National Library of Medicine (NIH) 2022. https://www.ncbi.nlm.nih.gov/
Gold JGI. Genomes Online Databases. Genomes Online Databases 2022. https://gold.jgi.doe.gov/simplesrch
KEGG. KEGG: Kyoto Encyclopedia of Genes and Genomes. Kyoto Encyclopedia of Genes and Genomes 2022.
European Genome Phenome Archive (EGA). EGA Consortium. What Is in the EGA? Studies in the EGA by Diseases 2022. https://ega-archive.org/
Leung YY, Kuksa PP, Amlie-Wolf A, Valladares O, Ungar LH, Kannan S, et al. DASHR: database of small human noncoding RNAs. Nucleic Acids Research 2016;44:D216–22. https://doi.org/10.1093/nar/gkv1188 DOI: https://doi.org/10.1093/nar/gkv1188
Human Cell Atlas. Human Cell Atlas. About Human Cell Atlas 2022. https://www.humancellatlas.org/
Rozenblatt-Rosen O, Stubbington MJT, Regev A, Teichmann SA. The Human Cell Atlas: from vision to reality. Nature 2017;550:451–3. https://doi.org/10.1038/550451a DOI: https://doi.org/10.1038/550451a
National Cancer Institute (NIH). Genomic data commons. The NCI’s Genomic Data Commons (GDC) 2022. https://gdc.cancer.gov/
Ríos Insuna D, Gómez Ullate Oteiza D. Big data. Conceptos, tecnologías y aplicaciones. vol. 1. 1st ed. Madrid, España: Consejo Superior de Investigaciones Científicas : Los Libros de la Catarata; 2019.
Thul PJ, Lindskog C. The human protein atlas: A spatial map of the human proteome. Protein Science 2018;27:233–44.
https://doi.org/10.1002/pro.3307 DOI: https://doi.org/10.1002/pro.3307
International Human Epigenetic Consortium (IHEC). Sharing epigenomes globally. Nature Methods 2018;15:151–151.
https://doi.org/10.1038/nmeth.4630 DOI: https://doi.org/10.1038/nmeth.4630
Stark C, Breitkreutz B-J, Chatr-aryamontri A, Boucher L, Oughtred R, Livstone MS, et al. The BioGRID Interaction Database: 2011 update. Nucleic Acids Research 2011;39:D698–704. DOI: https://doi.org/10.1093/nar/gkq1116
https://doi.org/10.1093/nar/gkq1116
Chatr-aryamontri A, Oughtred R, Boucher L, Rust J, Chang C, Kolas NK, et al. The BioGRID interaction database: 2017 update. Nucleic Acids Research 2017;45:D369–79. https://doi.org/10.1093/nar/gkw1102 DOI: https://doi.org/10.1093/nar/gkw1102
Nuño-Cabanes C, Ugidos M, Tarazona S, Martín-Expósito M, Ferrer A, Rodríguez-Navarro S, et al. A multi-omics dataset of heat-shock response in the yeast RNA binding protein Mip6. Scientific Data 2020;7:69. https://doi.org/10.1038/s41597-020-0412-z DOI: https://doi.org/10.1038/s41597-020-0412-z
ENCODE. The ENCODE (Encyclopedia of DNA Elements) project. Science (1979) 2004;306:636–40.
https://doi.org/10.1126/science.1105136 DOI: https://doi.org/10.1126/science.1105136
ENCODE. An integrated encyclopedia of DNA elements in the human genome. Nature 2012;489:57–74.
https://doi.org/10.1038/nature11247 DOI: https://doi.org/10.1038/nature11247
ENCODE. ENCODE Portal. ENCODE Stanford University 2022. https://www.encodeproject.org/
GENCODE. Statistics about the current GENCODE Release (version 40). GENCODE Human 2022.
https://www.gencodegenes.org/human/stats.html
Tuzun E, Sharp AJ, Bailey JA, Kaul R, Morrison VA, Pertz LM, et al. Fine-scale structural variation of the human genome. Nature Genetics 2005;37:727–32. https://doi.org/10.1038/ng1562 DOI: https://doi.org/10.1038/ng1562
Akiva P, Toporik A, Edelheit S, Peretz Y, Diber A, Shemesh R, et al. Transcription-mediated gene fusion in the human genome. Genome Research 2006;16:30–6. https://doi.org/10.1101/gr.4137606 DOI: https://doi.org/10.1101/gr.4137606
Nadeau JH, Topol EJ. The genetics of health. Nature Genetics 2006;38:1095–8. https://doi.org/10.1038/ng1006-1095 DOI: https://doi.org/10.1038/ng1006-1095
Amaral PP, Dinger ME, Mercer TR, Mattick JS. The Eukaryotic Genome as an RNA Machine. Science (1979) 2008;319:1787–9. https://doi.org/10.1126/science.1155472 DOI: https://doi.org/10.1126/science.1155472
Riddihough G, Zahn LM. What Is Epigenetics? Science (1979) 2010;330:611–611. https://doi.org/10.1126/science.330.6004.611 DOI: https://doi.org/10.1126/science.330.6004.611
Saletore Y, Meyer K, Korlach J, Vilfan ID, Jaffrey S, Mason CE. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology 2012;13:175. https://doi.org/10.1186/gb-2012-13-10-175 DOI: https://doi.org/10.1186/gb-2012-13-10-175
Liu N, Dai Q, Zheng G, He C, Parisien M, Pan T. N6-methyladenosine-dependent RNA structural switches regulate RNA–protein interactions. Nature 2015;518:560–4. https://doi.org/10.1038/nature14234 DOI: https://doi.org/10.1038/nature14234
Li X, Xiong X, Yi C. Epitranscriptome sequencing technologies: decoding RNA modifications. Nature Methods 2017;14:23–31. https://doi.org/10.1038/nmeth.4110 DOI: https://doi.org/10.1038/nmeth.4110
Zaccara S, Ries RJ, Jaffrey SR. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology 2019;20:608–24. https://doi.org/10.1038/s41580-019-0168-5 DOI: https://doi.org/10.1038/s41580-019-0168-5
Jiang C, Mithani A, Belfield EJ, Mott R, Hurst LD, Harberd NP. Environmentally responsive genome-wide accumulation of de novo Arabidopsis thaliana mutations and epimutations. Genome Research 2014;24:1821–9. https://doi.org/10.1101/gr.177659.114 DOI: https://doi.org/10.1101/gr.177659.114
Klosin A, Casas E, Hidalgo-Carcedo C, Vavouri T, Lehner B. Transgenerational transmission of environmental information in C. elegans. Science (1979) 2017;356:320–3. https://doi.org/10.1126/science.aah6412 DOI: https://doi.org/10.1126/science.aah6412
Chen L, Liu P, Evans TC, Ettwiller LM. DNA damage is a pervasive cause of sequencing errors, directly confounding variant identification. Science (1979) 2017;355:752–6. https://doi.org/10.1126/science.aai8690 DOI: https://doi.org/10.1126/science.aai8690
Zeraati M, Langley DB, Schofield P, Moye AL, Rouet R, Hughes WE, et al. I-motif DNA structures are formed in the nuclei of human cells. Nat Chem 2018;10:631–7. https://doi.org/10.1038/s41557-018-0046-3 DOI: https://doi.org/10.1038/s41557-018-0046-3
Schwartz W. Lynn Margulis, Origin of eukaryotic cells. evidence and research implications for a theory of the origin and evolution of microbial, plant, and animal cells on the Precambrian earth. XXII u. 349 S., 89 Abb., 49 Tab. New Haven-London 1970: Yale University. Zeitschrift Für Allgemeine Mikrobiologie 1973;13:186–186. https://doi.org/10.1002/jobm.19730130220 DOI: https://doi.org/10.1002/jobm.19730130220
Lolle SJ, Victor JL, Young JM, Pruitt RE. Genome-wide non-mendelian inheritance of extra-genomic information in Arabidopsis. Nature 2005;434:505–9. https://doi.org/10.1038/nature03380 DOI: https://doi.org/10.1038/nature03380
Walsh CT, Garneau-Tsodikova S, Gatto GJ. Protein Posttranslational Modifications: The Chemistry of Proteome Diversifications. Angewandte Chemie International Edition 2005;44:7342–72. https://doi.org/10.1002/anie.200501023 DOI: https://doi.org/10.1002/anie.200501023
Yang A, Cho K, Park H-S. Chemical biology approaches for studying posttranslational modifications. RNA Biology 2018;15:427–40. https://doi.org/10.1080/15476286.2017.1360468 DOI: https://doi.org/10.1080/15476286.2017.1360468
Harmel R, Fiedler D. Features and regulation of non-enzymatic post-translational modifications. Nature Chemical Biology 2018;14:244–52. https://doi.org/10.1038/nchembio.2575 DOI: https://doi.org/10.1038/nchembio.2575
Wang S-CM, Muscat GEO. Nuclear receptors and epigenetic signaling: Novel regulators of glycogen metabolism in skeletal muscle. IUBMB Life 2013;65:657–64. https://doi.org/10.1002/iub.1181 DOI: https://doi.org/10.1002/iub.1181
Agris PF, Narendran A, Sarachan K, Väre VYP, Eruysal E. The Importance of Being Modified, 2017, p. 1–50. DOI: https://doi.org/10.1016/bs.enz.2017.03.005
https://doi.org/10.1016/bs.enz.2017.03.005
Mohanta TK, Khan AL, Hashem A, Allah EFA, Yadav D, Al-Harrasi A. Genomic and evolutionary aspects of chloroplast tRNA in monocot plants. BMC Plant Biology 2019;19:39. https://doi.org/10.1186/s12870-018-1625-6 DOI: https://doi.org/10.1186/s12870-018-1625-6
Ribas de Pouplana L. The mitochondrial tRNA conundrum. Nature Reviews Molecular Cell Biology 2020;21:361–361.
https://doi.org/10.1038/s41580-020-0220-5 DOI: https://doi.org/10.1038/s41580-020-0220-5
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2022 Martínez Gómez P.

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-SinDerivadas 4.0.
Magna Scientia UCEVA proporciona un acceso abierto, libre y gratuito a su contenido, basado en el principio de que ofrecer al público un acceso libre a las investigaciones, ayuda a un mayor intercambio global del conocimiento. Lo cual, implica que los usuarios pueden leer, descargar, almacenar, imprimir, buscar, indexar y realizar enlaces a los textos completos de esta revista. Se permite distribuir los diversos artículos en las versiones post-print y oficial, sin previo permiso del autor o editor, considerando que el fin de este, no implica fines comerciales, ni la generación de obras derivadas; Solo se solicita la mención de la fuente así como la autoría. El titular del copyright será el o los autores que publiquen en Magna Scientia UCEVA.
Magna Scientia UCEVA está distribuida bajo los términos de la licencia https://creativecommons.org/licenses/by-nc-nd/4.0/deed.es